предметів
реферат
Протягом останніх 30 років ми провели багато базових досліджень на двох підривах Oryza sativa. indica, Zhenshan 97 (ZS97) та Minghui 63 (MH63). Щоб поліпшити роздільну здатність багатьох з цих досліджень, ми створили дві еталонні збірки еталонних геномів, використовуючи найсучасніші технології секвенування. Використовуючи технологію PacBio SMRT, ми створили понад 108 (ZS97) та 174 (MH63) Gb вихідних даних послідовності з 166 (ZS97) та 209 (MH63) груп клонів BAC та генерували
Дані послідовності 74 (MH63) Gb з парною кінцевою послідовністю у цілому геномі (WGS) з технологією секвенування Illumina. Отримавши ці дані, ми успішно склали два стандартні платинові стандарти, які були опубліковані. Ось повний набір вихідних даних, що використовуються для створення цих двох еталонних геномних збірок. Ці набори даних можуть бути використані для тестування нових програм для кращої збірки та анотації геномів, щоб допомогти відкрити нові точки зору на структуру, функції та еволюцію геному, а також допомогти забезпечити необхідну підтримку біологічних досліджень загалом.
Підсумок метаданих
Завантажте файл метаданих
Доступний до машини файл метаданих, що описує дані, що повідомляються (формат картки ISA)
Передумови та резюме
методи
Побудова бібліотеки BAC та послідовне секвенування
Дві бібліотеки BAC, що використовувались у цьому дослідженні, були раніше опубліковані 11. Коротко кажучи, частково перетравлену (тобто Hin dIII) та вибрану за розміром геномну ДНК від кожного сорту клонували у сайт Hin dIII pAGIBAC1 та трансформували у компетентні клітини T10 Escherichia coli DH10B. Обидві бібліотеки, названі OSIZBa (ZS97) та OSIABa (MH63), містили 36 864 клонів BAC, середні розміри вкладишів
125 кб (MH63) і були покриті
10, 7 × (MH63) кожного геному 11. Крім того, було створено 33, 969 (ZS97) та 35, 549 (MH63) двонаправлених кінцевих послідовностей BAC (BES) для першої половини кожної бібліотеки 11.
Фізичні карти
Фізичні карти з низьким охопленням (PM), описані вище за допомогою методу відбитків пальців SNaPshot, були описані вище11. Ми перетворили два ПМ, використовуючи метод профілювання цілого геному KeyGene (WGP) 12. WGP FingerPrint Contig (FPC) PM були побудовані в чотири етапи: (1) підготовка ДНК BAC, (2) підготовка плазмід BAC WGP з адаптерами для індексації та секвенування, (3) секвенування Illumina та (4) обробка біоінформатики. На кроці 4, використовуючи сценарії декодування WGP, було вилучено 99 996 (ZS97) та 103 609 (MH63) унікальних тегів WGP та позначено 32 829 (89,1%) та 30 749 (89,3%) BAC в бібліотеках ZS97 та MH63, відповідно. Використовуючи теги послідовності WGP для кожного клону BAC з кожної бібліотеки, було побудовано два нових ПМ із програмним забезпеченням FPC 13 (версія 9.4). Після ручного редагування та інтеграції з попередніми PMa SNaPshot покращені PM ZS97 та MH63 складалися з 539 та 401 контигів, що містять 28, 372 та 24, 519 клонів та 4, 457 та 6, 230 клонів як одиночні. Загальний розмір контейнерів оцінювався в 342 Мб для ZS97 (N50 = 940 кб) і 349 Мб для MH63 (N50 = 1270 кб).
Секвенування клону PacBio BAC
Клони BAC мінімального шляху прокладання (MTP) з кожного PM вибирались автоматично за допомогою спеціального сценарію та вручну розміщувались у посуді бібліотеки MTP з позначками OSIZBzz (ZS97) та OSIABzz (MH63) та зберігалися при -80 ° C. Загалом 4, 714 і Для ZS97 та MH63 було зібрано 4751 клонів BAC MTP. Повні списки клонів MTP наведені в Додатковій таблиці 1a-b.
Для секвенування клону PacBio BAC клони MTP BAC інокулювали в 96-лункові блоки росту глибинних лунок, вирощували протягом ночі при 37 ° C, центрифугували для осадження клітин і зберігали при -80 ° C до використання. Потім BAC були сформовані шляхом злиття свердловин із заморожених блоків в одну з шести конфігурацій: тобто асоціації одного корабля (12 BAC на пул), дві асоціації рядків (24 BAC на пул), чотири групи колон (32 BAC на пул), 6 груп колон (48 BAC на пул), вісім асоціацій стовпців (64 BAC на пул) або групи повного пансіону (96 BAC на пул). Потім ДНК екстрагували з кожної суміші, використовуючи стандартний протокол виділення плазмідної плазмідної лужної плазміди ДНК 14. Загалом було послідовно розподілено 166 (ZS97) та 209 (MH63) пулів (див. Нашу детальну схему об’єднання в Додатковій таблиці 2a-b). Використовуючи 16 мкг об’єднаної плазмідної ДНК, бібліотеки послідовностей PacBio готували згідно з наступними протоколами виробника, як описано для підготовки шаблону Blue Pippin на 20 кб. Послідовність SMRT проводили на приладі PacBio RSII з використанням хімії послідовностей P5/C3 та 3-годинних плівок.
Продукція сирого читання за допомогою PacBio
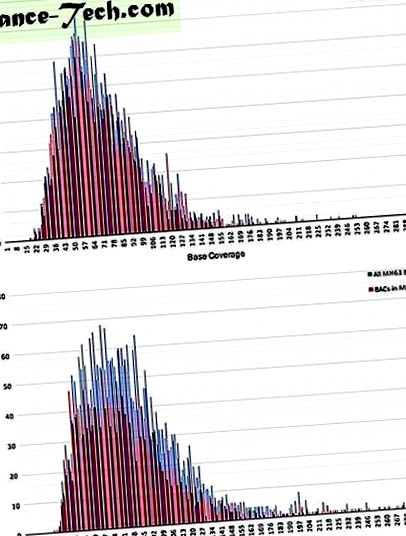
Аналіз підцепочок для обох груп послідовностей BAC ZS97 та MH63 проводили за допомогою порталу PacBio SMRT (версія 2.3.0). Для ZS97 дані з 227 комірок SMRT (які підраховують повторні реакції) були відокремлені та відфільтровані (тобто за допомогою протоколу RS_Subreads, мінімальна довжина зчитування полімерази = 50 bp, мінімальна якість зчитування полімерази = 75 і мінімальна довжина subread = 50 bp), що є результатом загальної кількості 107,5 Гб корисних даних послідовностей (загальна кількість реакцій полімерази = 11,6 М, кількість полімерази N50 = 12,8 кб; загальна кількість занурень = 17,7 М, середня довжина підшкірної шкіри = 5,7 кб, підчитана кількість N50 = 8,0 кб). Для MH63 дані з 317 комірок SMRT були оброблені, як зазначено вище (174 Гб корисних даних; 18,2 М полімерази зчитує, полімераза зчитує N50 = 12,1 кб; 26,8 М підчитування, середня довжина підчитування = 5,5 кб, підчитування N50 = 7, 8 кб).
Компіляція даних PacBio та ідентифікація послідовності BAC
Повнорозмірне зображення
Рослинний матеріал, побудова бібліотеки ДНК для послідовного висвітлення
Ми також використовували технологію послідовного короткого читання Illumin для секвенування геномів ZS97 та MH63, використовуючи підхід цілого геномного дробовика (WGS). Рослинні матеріали вирощували в теплиці, а листя 4 тижня використовували для вилучення геномної ДНК за допомогою стандартних процедур. Парні бібліотеки, включаючи невеликі вставки (
300 bp) та дві бібліотеки великих вкладишів (5 kb, 10 kb) були підготовлені з парними кінцевими та парними наборами Illumina (таблиця 1). Щонайменше 5 мкг геномної ДНК було фрагментовано шляхом розпилення стисненим газом азоту для кінцевих бібліотек з короткою вставкою. Більша кількість високоякісної геномної ДНК (10-30 мкг) була потрібна для побудови бібліотеки з довгою вставкою пари пари. Бібліотеки послідовностей Illumina були підготовлені відповідно до протоколу виробника. Бібліотеки були послідовно розподілені на Illumina HiSeq 2000 відповідно до стандартних протоколів Ilulina (Illumina, Сан-Дієго, Каліфорнія). Загальна кількість даних про необроблену послідовність, що генерується для кожного сорту, становила
97, 5 Гб даних для ZS97 та
74,0 Гб даних для MH63. Після ряду етапів фільтрації даних вони були використані для видалення штучних показань, спричинених дублюванням ПЛР та забрудненням адаптерів, загалом 87,4 Гб високоякісних зчитувань (> 200 ×) для ZS97 та
Для MH63 було отримано 67,9 Гб (> 170 ×) (табл. 1). Якість бібліотеки перевіряли шляхом визначення розподілу розмірів вкладишів та глибини послідовності. Фактичну довжину вставки визначали шляхом зіставлення спарених кінців з O. sativa subsp. японіка cv. Довідковий геном Nipponbare (Nipponbare RefSeq) 17 .
Стіл в натуральну величину
Illumina читає попередню обробку та збірку de novo
Ми використали новий гібридний підхід, що поєднує de novo складання та еталонні методи 18 для складання показань Illumina для кожного геному. Всі послідовні показання з ZS97 та MH63 були виправлені за допомогою Medusa 19 та Quake 20. Виправлені зчитування були усічені на їх кінцях, якщо якість їх послідовності була менше 20 за допомогою fastx_tool_kit (//hannonlab.cshl.edu/fastx_toolkit/index.html), а лічильники були видалені за допомогою cutadpat 21 (//github.com/) Marcely/cutadapt /). Потім оброблені дані були зіставлені в Nipponbare RefSeq за допомогою BWA 22. Усі показання, які відображались у суцільній зоні, були взяті окремо, а суміжну зону покриття визначали як блок. Визначення блоків та суперблоків є однаковим, як описано вище 23, за винятком того, що наша мінімальна довжина суперблоку становила 20 кб, а перекриття суперблоку - 2 кб. Локально ми de novo зібрали всі показання, зібрані в суперблоці, використовуючи SOAPdenovo 24. Випробовували низку різних к-мерів, а решту contigs з найвищими значеннями N50 зберігали. Отримані контиги збирали з AMOS 25 з використанням відповідних еталонних хромосом як напрямних. Некартовані зчитування були зіставлені з геномом indica 9311 та зібрані за тією ж процедурою.
Потім Nucmer 27 був використаний для вирівнювання всіх суперконтролів до Nipponbare RefSeq. Потім ми перевірили глибину охоплення та відображення розривів між сусідніми районами. Зіставлені зчитування були обрані шляхом збільшення мостових прогалин у геномах MH63 та ZS97 на 200 п.н. з обох сторін кожної прогалини. Усі вибрані числа в цих областях були складені з використанням cap3 28. Зібрані контиги були суміщені з двома безперервними суперконтролями і з'єднані на основі їх послідовного розташування.
Для включення специфічних послідовностей MH63 та ZS97, яких не було в геномах Nipponbare та 9311, ми виконали повну збірку геному de novo, використовуючи всі оброблені показання SOAPden 24, а потім вирівняли збірки de novo, зібрані з комбінованими надпровідниками, та додатково перев’язали відповідні суперконтролі . Нарешті, риштування виконували за допомогою SSPACE 29, а прогалини заповнювали gapCloser (//sourceforge.net/projects/soapdenovo2/files/GapCloser/).
Остаточна статистика наборів WGS Illumina ZS97 та MH63 наведена в таблицях 2 та 3 і була використана для заповнення прогалин між сусідніми контигентами PacBio.
Стіл в натуральну величину
Стіл в натуральну величину
Побудова псевдомолекул у два етапи
На першому кроці всі дані послідовності BAC були введені в Master Puzzle Master 16 (GPM) для створення контигів послідовностей на основі PacBio за допомогою фізичних карт та довідкових посібників Nipponbare RefSeq 17. GPM - це напівавтоматизований конвеєр, розроблений для інтеграції даних логічних взаємозв’язків (наприклад, фізичних карт) у послідовності ешафотів у послідовності хромосомного діапазону. Як результат, 318 (ZS97) та 216 (MH63) зібрані контиги були влаштовані та орієнтовані, а також закріплені у відповідних хромосомах після ручного огляду, редагування та видалення надлишків. Остаточні збірки на основі PacBio складалися з послідовностей з 3862 (ZS97, включаючи 57 не-MTP) та 3254 (MH63, включаючи 77 не-MTP) унікальних BAC.
Оскільки в цьому дослідженні ми використовували стратегію секвенування на основі фізичних карт, прогалини в наших фізичних картах WGP є основними причинами розривів у збірках на основі PacBio. Отже, другим кроком було заповнення прогалин шляхом інтеграції даних збірки Illumina WGS. Однак, щоб мінімізувати вплив потенційно неправильних даних у даних Illumina, ми використовували лише contra Illumina, який на цьому етапі міг повністю з'єднати два сусідні контакти PacBio. Загалом 81 пробіл у ZS97 (8 988 328 п.н.) та 35 у MH63 (3 127 191 п.н.) заповнили 76 (ZS97) та 35 (MH63) послідовностей послідовності Illumina (Додаткова таблиця 4a-b). В результаті остаточні гібридні псевдомолекули (ZS97RS1 та MH63RS1) містили 237 (включаючи 2 незакриті) та 181 (включаючи 2 непідтверджені) контиги для ZS97 та MH63.
Записи даних
Усі непослідовні дані доступні у сховищі даних iPlant (Таблиця 4). Бібліотеки OSIZBa та OSIABa BAC, або окремі клони BAC, можна отримати в Ресурсному центрі BAC/EST AGI за адресою www.genome.arizona.edu/orders. Кінцеві послідовності BAC раніше зберігались у GenBank під номерами приєднання KG737749 - KG771717 (ZS97, посилання на дані 1: GenBank KG737749 - KG771717) та KG702200 - KG737748 (MH63, цитування даних 2: GenBank KG702200 - KG737748).
Стіл в натуральну величину
Вихідні дані послідовності PacBio доступні в архіві короткого читання NCBI (SRA) під номерами приєднання SRP071597 (ZS97, Посилання на дані 3: Архів читання послідовності NCBI SRP071597) та SRP071598 (MH63, Посилання на дані 4: Архів читання послідовності NCBI SRP071598). Всі дані послідовності Illumina можна знайти під номером приєднання SRP071944 (ZS97 та MH63, Цитування даних 5: Архів архівів читання послідовності NCBI SRP071944). Через несподівану помилку на диску ми втратили вихідні дані послідовності PacBio з 57 пулів. На щастя, всі прогони HGAP були заархівовані в сховищі даних iPlant під розділом «завдання смерті», де можна було отримати відфільтровані підшари цих відповідних груп. Зібрані дані від Illumina доступні для збірки NCBI під номерами приєднання GCA_001618795 (ZS97, посилання на дані 6: Збір NCBI GCA_001618795) та GCA_001618785 (MH63, посилання на дані 7: Збір NCBI GCA_001618785).
Остаточні псевдомолекули геному (версія 1) для кожного еталонного геному зберігались у збірці NCBI під номерами приєднання GCA_001623345 (ZS97RS1, посилання на дані 8: Збірка NCBI GCA_001623345) та GCA_001623365 (згадане посилання 9: Збірка NCBI_G0065).
Технічна перевірка
По суті, кожна геномна еквівалентна бібліотека BAC була свіжо вирощена в скопійованих наборах з 384-лункових планшетів, і тривимірне об'єднання було проведено на бактеріальних клітинах з подальшим ростом клітин та екстракцією плазмідної ДНК за допомогою хімічного лужного лізису. Збірки ДНК розщеплювали рестрикційними ферментами (EcoRI/MseI), після чого проводили лігування олігомерів розміром у пул, які були розроблені для конкретної локалізації адрес клонів BAC та асоціювання з послідовностями. Після ампліфікації змішаних молекул було проведено секвенування Illumina, і отримані дані були проаналізовані, щоб ідентифікувати позначку послідовності 50 п.н. для кожної конкретної адреси клону BAC та сформувати набори смуг як вхідні дані до FPC. FPC запускався в умовах високої жорсткості (HS): спочатку з 'допуском = 0 [фіксований], відсіканням = 1e-15', потім вибравши DQ (у 3 кроки: Cutoff = 1e-18, 1e-21, 1e -24 ) був використаний для розповсюдження проблемних контигів. Після створення результуючих ПС HS, ми виконали етап злиття від кінця до кінця (Cutoff = 1e-9) і включили мічені одиночні елементи в контиги (Cutoff = 1e-12), щоб створити карти обмеженої жорсткості (RS). Карти WGP RS були модифіковані вручну шляхом інтеграції з попереднім низьким покриттям PMs 11 .
На кроці GPM 'AssemblyRun' для створення послідовностей послідовностей на основі BAC параметри за замовчуванням для злиття двох послідовностей BAC були 'minOverlapSeqToSeq = 1000 bp' та 'identitySeqToSeq = 99%', з перекриттями, необхідними в кінці обох послідовностей. Ми використовували Nipponbare RefSeq 17 як посилання для призначення номерів хромосом для складання контигу, а також для їх упорядкування та орієнтування. Крім того, лише одна копія надлишкової послідовності перекриття була збережена в зібраному контигу, без надання переваги визначенню того, який фрагмент послідовності BAC був збережений. Однак послідовності без пропусків мали вищий пріоритет, ніж прогалини. Всі контиги були перевірені вручну та скориговані за необхідності за допомогою браузера GPM 16. Коли ми використовували зібрані контиги Illumina, щоб заповнити прогалини між двома контигами на основі BAC, ми вибрали лише контиги Illumin, які могли повністю з'єднати два сусідні контиги на основі BAC і, що важливо, такі перекриття ('minOverlapSeqToSeq = 1000 bp' a 'identitySeqToSeq = 99% ") має відбуватися в кінці обох контигів." Коли в цих регіонах знаходили надлишкові сайти, послідовності послідовностей на основі BAC завжди зберігалися в кінцевих збірках геномів.
Цей документ є першим виданням вихідних даних для збирання геномів рису Zica ZS97 та MH63, а також містить перші версії двох наборів високоякісних псевдомолекул в науковому співтоваристві. Технології секвенування ДНК та програми складання послідовностей швидко змінюються, і представлені тут набори даних містять кілька типів секвенування, які можна використовувати для розробки нових методологій та програмних засобів як тестових входів.
Детальніше
Як цитувати цю статтю: Zhang, J. та співавт. Генерування двох еталонних геномів рису індика з даними тривалого зчитування PacBio та секвенуванням Illumina. Наук. Дані 3: 160076 doi: 10.1038/sdata.2016.76 (2016).
Цитування даних
GenBank KG737749 - KG771717 (2013)
GenBank KG702200 - KG737748 (2013)
Архів для читання NCBI SRP071597 (2016)
Архів читання послідовності NCBI SRP071598 (2016)
Архів читання послідовності NCBI SRP071944 (2016)
- Закоханий у Томаша Клюса, через два тижні він знав, що хоче дитину від Тамари
- Зупинити втрату біорізноманіття та створити здорову та стійку систему харчування у Європі
- Оздоровлення в готелі Фонтана в Брно з напівпансіоном для двох та дитини до 6 років безкоштовно, Чехія - Брно
- Yelp використовує машинне навчання, щоб створити список улюблених страв
- З двох вбивств може бути чотири