предметів
реферат
Матеріали і методи
Етичне твердження
Усі роботи на тваринах, описані в цьому дослідженні, були схвалені Інституційним комітетом з догляду та використання тварин (IACUC) при Університеті Арканзасу, і всі експерименти проводились відповідно до затверджених керівних принципів та норм.
Дизайн експерименту з тваринами
Збір крові та вилучення ДНК
Зразки крові відбирали у 5, здавалося б, здорових птахів на загон 14, 41 та 49 днів для аналізу мікроорганізмів, щоб отримати 240 зразків крові (5 птахів на загон × 16 загонів на вік відбору проб x 3 вікові категорії). Ще 12 зразків крові відібрали у птахів, яким піддавали BCO (n = 12; 5 і 7 зразків віком 41 та 49 днів, відповідно), що дозволило проаналізувати загалом 252 зразки для профілювання 16S рРНК. Зразки крові брали асептично з вени крила за допомогою вакутейнерів EDTA. Один мл зразка крові центрифугували (5000 об/хв протягом 5 хвилин при кімнатній температурі) з використанням мікроцентрифуги, а 200 мкл бафі-шару збирали в стерильній ламінарній камері і зберігали при -20 ° C. Геномну ДНК витягували з бафі-шарів за допомогою BiOstic® Bacteremia DNA Isolation Kit (MoBio) відповідно до інструкцій виробника. Зразки ДНК аналізували за допомогою флуорометра Qubit 2.0 (Life Technologies) щодо кількості та чистоти та зберігали при -20 ° C.
Протокол ПЛР для ампліфікації гена 16S рРНК
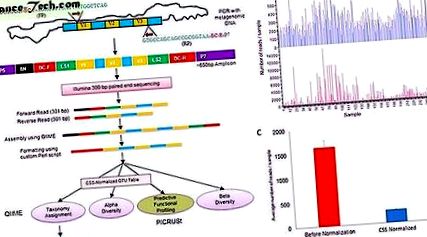
( a ) Блок-схема, що представляє підготовку бібліотеки, форматування показань, прийнятних для аналізу QIIME, які далі аналізували за допомогою LefSe та PICRUSt. * Для аналізу PICRUST була використана стандартизована таблиця OTU. (FP = прямий праймер, RP = зворотний праймер, V = варіативна область гена 16s РНК, P5 і P7 = праймери послідовності Illumina, N = випадковий нуклеотид, BC-F = штрих-код прямого праймера, BC-R = зворотний штрих-код праймера) і LS = послідовність лінкера). ( b ) Гістограма, що показує розподіл 252 зразків до (червоний) та після нормалізації (синій) таблиці OTU. c ) Гістограма, що показує середню кількість показань на вибірку зі стандартною похибкою до (червоний) та після нормалізації (синій).
Повнорозмірне зображення
Аналіз даних
Блок-схема, що описує аналіз послідовності 16S рРНК, показана на фіг. 1а. Зчитування парного кінця парного кінця з послідовності Illumin вперед та назад було складено, використовуючи кількісні уявлення про екологію мікроорганізмів, QIIME 1.9.1 25, за допомогою скрипта join_paired_ends.py за допомогою методу fastq-join. Потім послідовності штрих-коду прямого зчитування (BC-F) та зворотного зчитування (BC-R) були об'єднані та приєднані (5 'кінець) до зібраної послідовності (3' кінець) після видалення 8 випадкових послідовностей нуклеотидів та 27F та Послідовності праймерів 533R за власним сценарієм Perl, який створив файл fastq, сумісний з подальшим аналізом за допомогою QIIME.
Зчитування групували за допомогою UCLUST 26, а оперативні таксономічні одиниці (OTU) з можливістю закриття посилання проводили за допомогою видання 13_8 бази даних GreenGenes. Таблиця OTU BIOM (Biological Observation Matrix) була нормалізована до QIIME (normalize_table.py) за допомогою методу сукупної шкали (CSS), який потім використовувався для таксономічних присвоєнь, оцінки альфа-різноманітності, ідентифікації біомаркерів та прогнозування вмісту функціональних метагеном. Індекси альфа-різноманітності порівнювали за допомогою одностороннього дисперсійного аналізу (ANOVA), а пост-хок-аналіз проводили методом HSD Tukey-Kramer. Аналіз схожості (ANOSIM) між групами метаданих проводили за допомогою незваженої метрики відстані UniFrac із QIIME (compare_categories.py). Крім того, для оцінки бета-розмаїття за допомогою QIIME (24) були проведені головні координаційні аналізи (PCoA) з використанням зважених та незважених метрик UniFrac на різних глибинах вибірки.
Ідентифікацію біомаркерів проводили з використанням величини ефекту лінійного дискримінантного аналізу (LEfSe) 27. Метагенний функціональний вміст мікробіомів курячої крові прогнозували за допомогою PICRUSt (філогенетичне дослідження спільнот шляхом реконструкції ненаблюдених станів) з використанням послідовностей генів 16S рРНК 28. Бактеріальну мережу візуалізували за допомогою Cytoscape 3.2.1.
результат
Демультиплексування QIIME та якісна фільтрація дозволили отримати 4, 153, 965 зібраних послідовностей в діапазоні від 40 до 580 п.н. із середньою довжиною 469 п.н., що свідчить про наявність химерних послідовностей внаслідок аберативного посилення ПЛР або невідповідності. У цьому дослідженні ми використовували закритий еталонний метод OTU, оскільки досліджуємо новий тип мікробіомів, для яких структура громади, різноманітність та членство в основному невідомі. Однак ми також порівняли результати, отримані з використанням закритого еталонного методу, з відкритими еталонними методами та методами відбору OTU de novo (Додаткова таблиця 2). Хоча відкрите посилання та метод відбору OTU de novo призвели до більшої кількості OTU (у середньому 196 та 218 відповідно), а також до вищих значень на вибірці, були зафіксовані значні пропорції (в середньому 72, 5 та 79 відповідно%). ), які не були включені до жодної таксономічної групи в поточній базі даних GreenGenes 13_8. На додаток до ефективного видалення всіх шумових або химерних послідовностей у подальшому аналізі, ми віддали перевагу закритій збірці OTU-колекції перед двома іншими методами.
Закрита довідкова таблиця OTU мала середню глибину вибірки 1621 для кожного зразка (± 139, 07), як показано на фіг. Таблиця OTU нормалізувалася за допомогою сукупного масштабування (CSS) за допомогою QIIME (рис. 1b, c). Хоча середні значення зразків були зменшені внаслідок нормалізації більш ніж у 6 разів (266, 55 на зразок), вони також зменшили стандартну похибку (± 7, 01). Нормована таблиця OTU CSS була використана для призначення таксономії, оцінок альфа-різноманітності, аналізу LEfSe та аналізу PICRUt. Аналіз бета-різноманітності проводили на різних глибинах дискретизації та ANOSIM із 999 перестановками, використовуючи незважену метрику UniFrac при однаковій глибині дискретизації 400 кадрів на зразок.
Присвоєння таксономії
a ) Гістограма на рівні стовбура з діаметром. Різні літери над кожним стовпцем вказують на суттєві відмінності (ANOVA, тест Тукі-Крамера, с.4 .
Ящик паркан експериментальної категорії з пультом. Поля показують середній, 25-й та 75-й процентилі із значеннями поза нижньою та верхньою крайностями. Коробки з різними літерами суттєво відрізняються (стор
a ) Ділянка PCoA здорових (n = 171) проти BCO (n = 9) курей з використанням зважених показників UniFrac при однаковій глибині відбору проби 400 зразків. ( b ) Ділянка PCoA 14 днів (n = 30), 41 день (n = 41) проти 49 днів (n = 36) старих курей із зваженими показниками UniFrac при однаковій глибині вибірки 1000 кадрів на зразок. ( c ) Діаграма PCoA стартерів (n = 67) проти курців-фінішерів (n = 154), яких годували дієтами з використанням незваженої метрики UniFrac при однаковій глибині відбору проб 200 кадрів на зразок.
Повнорозмірне зображення
Істотну різницю в бактеріальних спільнотах здорових та ВСО курей також було проілюстровано ієрархічною кластеризацією. Курей BCO з 49-денними курчатами відокремлювали від здорових курчат. Однак у 41-денних курей BCO не було виявлено ознак значної кластеризації (рис. 5а). Мережевий аналіз між курами (252 зразки) та OTU (види бактерій) показав деякий ступінь роздільної здатності у моделях взаємодії між BCO та здоровими курями (рис. 5b), вказуючи на різні бактеріальні спільноти.
a ) Ієрархічне групування здорових та ВСО курей на основі мікробної крові. Філогенетичне дерево було створено за допомогою програмного забезпечення FigTree V1.3.1 із зваженими метриками UniFrac з використанням даних попередньої нормалізації. Зелені та червоні лінії представляють курей BCO протягом 41 та 49 днів. Чорні лінії - здорові кури. Цифра в кінці рядка показує ідентифікатор зразка курки. b ) Бактеріальна мережа мікроорганізмів курячої крові, вироблених за допомогою Cytoscape V3.2.1. Червоні, сині та білі вузли представляють курей BCO, здорових курей та OTU. Зелена облямівка - це куряча мережа BCO, а помаранчевий край у здорових курей.
Повнорозмірне зображення
Ми відзначили, що всі птахи BCO були лише з двох вікових груп (день 41 та 49) та двох груп (W35-56 та W1-56). Тому ми провели додатковий аналіз бета-різноманітності, використовуючи підмножину з 77 зразків, що належать до цих вікових груп та до груп на основі губ, включаючи 65 здорових птахів та 12 птахів BCO. Аналіз ANOSIM на основі зважених показників UniFrac показав більший поділ між BCO та здоровими курами (R = 0,5293, p = 0,001) порівняно із загальним набором даних (n = 252). Графік PCoA, показаний на фіг. 6 також підтримує результат аналізу ANOSIM.
Ділянка PCoA здорових (n = 51) проти BCO (n = 9) курей з використанням зважених показників UniFrac при однаковій глибині відбору проби 400 зразків на зразок.
Повнорозмірне зображення
Біомаркери BCO
Таксономічні групи, які за чисельністю відрізняються між здоровими курями та курями BCO, були ідентифіковані з використанням величини ефекту лінійного дискримінантного аналізу (LEfSe) з = 0,05, оцінка LDA щонайменше 2 та відносна кількість вище 0,1. Загалом 26 функцій мали суттєво відрізняється кількість між здоровими та BCO курями. На рівні роду мікробіоди крові ВСО курчат диференційовано збагачувались стафілококами, гранулікателлами та мікробактеріями, тоді як здорові кури збагачувались псевдомонадою, енгідробактером та аквабактерією (рис. 7б). Ми також виявили, що штам Firmicutes збагачений курями BCO. Подібним чином альфапротеобактерії були високо збагачені у курей ВСО, тоді як бетапротеобактерії та гаммапротеобактерії у здорових курей на рівні класу (рис. 7б).
a ) Таксономічна кладограма, зроблена на основі аналізу LEfSe. Червоний та зелений містять таксони, збагачені BCO та здорових курей. Яскравість пропорційна кількості таксону. ( b ) Податки, збагачені курями BCO, показані червоним кольором з від'ємним балом LDA, а здорові кури в зеленому стані з позитивним балом LDA (> 3, 5 в обох випадках). Таксон між двома підкресленнями пропонується як назва бази даних GreenGene.
Повнорозмірне зображення
Передбачувана функціональна генетична здатність мікроорганізмів курячої крові
( a ) Відносна кількість рівнів КЕГ COG на рівні 1 у мікробіомах курячої крові. ( b ) Відносна кількість (≥ 0, 5) шляху KEGG на рівні 3 функціональних шляхів, створених програмним забезпеченням JMP. ( c ) диференційовані символи (категорія KEGG COG, відносна кількість ≥ 0, 5), отримані за допомогою LEfSe з оцінкою LDA ≥ 2, 8.
Повнорозмірне зображення
обговорення
Це перше всебічне дослідження для аналізу бактеріальних мікробів, що існують у крові хребетних, які не є людиною. В останні роки зростає інтерес до характеристики мікробіону, пов’язаного з різними тканинами організму за різних умов здоров’я. Частини тіла, які вважаються стерильними, такі як кров 30, шлунок 31, сечовий міхур 32, легені 33, кістки, суглоби 4 та груди 34, мають свій початковий мікроорганізм. Дисбіоз мікробіозу тканин асоціюється з різними захворюваннями, включаючи серцево-судинні захворювання 10, цукровий діабет 35, неалкогольну жирову хворобу печінки (NASHD) 36, запальну хворобу кишечника (IBD) 37, псоріаз 38, ожиріння 39, астму 39 в дитячому віці, функціональне захворювання кишечника 40 та колоректальний рак 41. Тут ми вивчили мікробіоз курячої крові, щоб виявити потенційні бактеріальні біомаркери, пов’язані з BCO.
Результат аналізу даних у цьому дослідженні показав існування бактеріальних спільнот, що містять від 30 до 40 ОТУ в крові курчат-бройлерів, незалежно від віку та інших умов навколишнього середовища або господаря. Аналіз бета-різноманітності (рис. 4а та 6), ієрархічний кластерний аналіз (рис. 5а) та аналіз бактеріальних мереж (рис. 5б) на основі мікробної крові та ієрархічна кластеризація на основі прогнозованого метагенома мікробіонів крові (рис. 8б) свідчать про те, що бактеріальні спільноти У крові птахів BCO відрізняються від спільнот здорових птахів, що вказує на наявність певного селективного тиску, що сприяє зміщенню мікробіомерів крові у птахів BCO. Однак лише все дерево PD показало значну різницю в альфа-різноманітності між BCO та здоровими птахами, що узгоджується з філогенетично віддаленим мікробіозом крові у BCO птахів, як показано в аналізі бета-різноманітності (рис. 4а та 6).
У нашому неопублікованому супутньому дослідженні зразки крові з тієї ж зграї безпосередньо посівали на багаті агарові середовища. Результат показав, що кількість колоній на 49 день була постійно вищою на дротяних підлогах (L35W і W56) порівняно з підстилкою (L56), що свідчить про те, що напруга на дротяній підлозі сприяла бактеріємії, ймовірно, через транслокацію бактерій через епітелій кишечника. Крім того, кількість колоній на 49 день також була стабільно вищою у кульгавих птахів порівняно зі здоровими птахами. Ці результати вказують на сильну кореляцію між напругою дротяної підлоги, ступенем тяжкості бактеріємії та кульгавістю BCO.
Одним із практичних застосувань цього дослідження було виявлення бактеріальних біомаркерів, які можна було б використовувати для ідентифікації окремих курчат-бройлерів у більш ранньому віці, які сприйнятливі до розвитку ВСО у більш пізньому віці. Дійсно, аналіз даних у цьому дослідженні виявив таксономічні групи на різних рівнях, які значно збагачені у птахів BCO порівняно зі здоровими птахами. Цікаво, що рід Staphylococcus є однією з 18 ознак (включаючи 3 роди), значно збагачених у зразках BCO, підкреслюючи важливість цього роду, який часто був виділений із уражень BCO 3, 4, 5. Нещодавно Аль-Рубай та ін. 44 зазначив, що виклик бройлерів з S. agnetis, який у своєму дослідженні був найчастіше виділеним стафілокозним видом із уражень BCO, значно збільшив травму з 10 (контроль) до 40%, тоді як виклик іншим ізолятом Enterococcus faecalis зменшив захворюваність на опорно-руховий апарат, Це може свідчити про потенційне значення S. agnetis як збудника BCO у курчат-бройлерів, хоча наші дані профілювання гена 16S рРНК не дають значущої інформації про види Staphylococcus через обмежену таксономічну роздільну здатність.
Однак у цьому дослідженні BCO було виявлено лише у птахів у віці 41 та 49 днів, і тому бактеріальні біомаркери мають обмежене значення для ранньої діагностики BCO-чутливих птахів. Цей аспект слід ретельно розглянути в експериментальній схемі для майбутніх досліджень, щоб дозволити ідентифікувати потенційні бактеріальні біомаркери у зразках крові молодих птахів, які передбачають розвиток ВСО у старших вікових групах.
Мікроби крові, проаналізовані в цьому дослідженні, мають значний вплив на стан здоров’я курчат-бройлерів, включаючи патогенез ВСО, як продемонстровано в цьому дослідженні, а також на інші захворювання або стресові стани у курчат-бройлерів.
Детальніше
Як цитувати цю статтю: Mandal, RK et al. Дослідження мікробної крові та її потенційний зв’язок з бактеріальним хондронекрозом та остеомієлітом (BCO) у бройлерів. Наук. Респ. 6, 25882; doi: 10.1038/srep25882 (2016).