Предмети
Резюме
Мало вивчений пікорнавірус, людський пареховірус 3 (HPeV3) спричиняє сепсис новонароджених без наявної терапії. Наша структура роздільної здатності 4,3 Å для HPeV3 сама по собі та роздільна здатність 15 Å у комплексах з моноклональними антитілами людини Fab демонструє очікувану структуру капсиду пікорнавірусу з трьома різними характеристиками. По-перше, 25% геному РНК HPeV3 на 60 ділянках є впорядкованим, що підтверджується асиметричною реконструкцією, і взаємодіє із збереженими областями білків капсидів VP1 та VP3. По-друге, термін VP0 N стабілізує внутрішню поверхню капсиду, на відміну від інших пікорнавірусів, у яких при викиді, як VP4, він утворює канал транслокації РНК. Нарешті, гідрофобна кишеня VP1, місце зв’язування антипікорнавірусного препарату, плеконарилу, блокується і, отже, непридатна для розвитку вірусів. У сукупності ці результати пропонують напрямок розвитку нейтралізуючих антитіл, противірусних препаратів, заснованих на РНК-білкових взаємодіях, та дисекції збірки вірусів на основі зародження РНК.
Вступ
Picornaviridae - це сімейство маленьких позитивно-чуттєвих ікосаедричних моделювальних одноцепочечних РНК-вірусів. Пареховірус А є видом у цій родині з 16 генотипами і в основному асоціюється з легкими інфекціями у людей, особливо у дітей. Однак новий патоген, пареховірус людини 3 (HPeV3), може спричинити серйозні інфекції центральної нервової системи, такі як менінгіт 1, і є основною причиною неонатального сепсису 2. Немає доступних противірусних препаратів та вакцин для боротьби з інфекцією HPeV. На відміну від багатьох інших пікорнавірусів, HPeV погано характеризуються як за структурою, так і за функцією, за винятком HPeV1, де рецептор 3 відомий. Великі відмінності в тропізмі, виявлені HPeV3, порівняно з іншими HPeV, роблять важливим дослідити структурні властивості HPeV3 для кращого розуміння його патогенезу та можливого зв'язування з рецепторами.
Ми використовуємо кріоелектронну мікроскопію та реконструкцію зображень для аналізу структури HPeV3 як окремо, так і в комплексі з моноклональними антитілами людини Fab. Структура віріону показує, що зв'язуючі кишенькові ліки VP1, такі як плеконарил, навряд чи будуть зв'язуватися з HPeV; що VP0 є важливим білком для стабілізації внутрішньої поверхні капсиду і, нарешті, що збірка HPeV, ймовірно, контролюється за рахунок багаторазових взаємодій геному з капсидом через збережені амінокислоти в VP1 і VP3 та структури стовбура та петлі в РНК. Ми виділили та охарактеризували моноклональне антитіло людини, специфічне для HPeV3, яке може бути дуже корисним для прогресування діагностики вірусів та вивчення взаємодії вірус-господар.
Результати і обговорення
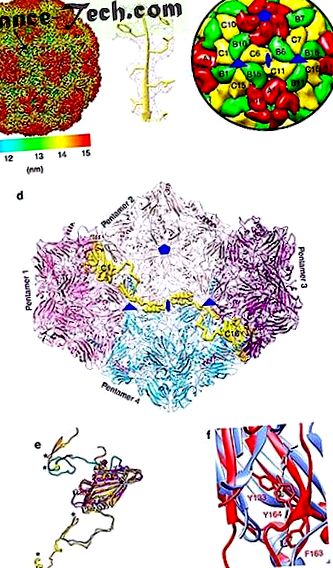
Будова HPeV3
Повнорозмірне зображення
Впорядкована структура РНК HPeV3
Повнорозмірне зображення
Повнорозмірне зображення
Гідрофобний кишеньковий VP1
Противірусний препарат, плеконарил та його похідні загалом блокують ентеровірусні інфекції, такі як EV-D68 (посилання 4). Ці препарати виганяють ліпід ("кишеньковий фактор"), присутній у гідрофобній кишені β-бочки VP1, і блокують РНК-покриття, стабілізуючи капсид 4. Канал до гідрофобної кишені в HPeV3 VP1 перекритий трьома великими консервативними бічними ланцюгами Y133, F163 та Y164 (рис. 1f та 3) порівняно з EV-D68 (посилання 4). Тому ми можемо пояснити спостереження, що плеконаріл не працює в клініці 2, і передбачити, що такі імітатори кишенькових факторів не будуть працювати проти будь-якого з послідовних до цього ВПЕВ.
HPeV3 - Fab складна структура
Ми виділили специфічне для HPeV3 моноклональне антитіло людини AT12-015, яке зв’язувалось з ізолятами HPeV3 152037, A308/99 та двома клінічними ізолятами в інфікованих клітинах. Він не нейтралізував 152037 в аналізі на основі клітинних ліній Vero. Структура ізоляту HPeV3 152037 була розв’язана в комплексі з Fab AT12-015 (Додаткова таблиця 1; Рис. 4а; Додаткова Рис. 1). Молекули Fab розпізнають конформаційний епітоп на краю бочки (рис. 4а). Вклад надходить із областей як у VP3 (петлі hZβB, βChA, βEβF та βGβH), так і у VP1 (βBβC, βCβD та βEβF, C), що зберігається переважно у HPeV3, що пояснює специфічність антитіла до цього генотипу (рис.3). . Відбиток пальця Fab охоплює термінал VP1 C, де багато інших HPeV містять мотив RGD для зв'язування з їх рецепторами інтегрину (рис. 3 та 4b) 3. Оскільки AT12-015 є специфічним для HPeV3 і не зв’язується з жодним з інших тестованих генотипів HPeV, він може бути дуже корисним для просування діагностики вірусів та вивчення взаємодій вірусу та господаря. Подальше порівняння з епітопами нейтралізуючих сироваток пацієнта може допомогти нам зрозуміти механізм нейтралізації у пацієнтів 13 .
( до ) Радіальне кольорове зображення поверхні поверхні комплексу HPeV3-Fab AT12-015 при роздільній здатності 15 Å, показаному при 1,5σ вище середнього порогу. Молекули Fab (червоні) зв'язуються навколо району каньйону. ( b ) Дорожня карта HPeV3. Слід Fab (червоний контур) відображається на VP3 (зелений) і VP1 (червоний).
Повнорозмірне зображення
Загалом, ця робота демонструє множинні стабілізуючі взаємодії РНК-капсиди як нову мішень для пареховірусних препаратів. Ми виявляємо незвичну стабільність цих капсидів на внутрішньопентамерному рівні за допомогою взаємодій VP3 та на інтерпентамері через VP0, і ми припускаємо, що механізм покриття геному HPeV буде значно відрізнятися від інших пікорнавірусів.
Методи
Очищення та інактивація вірусів.
HPeV1-Harris та HPeV2-2008 були надані Нідерландським національним інститутом охорони здоров’я та навколишнього середовища, Білтховен, Нідерланди. HPeV4-251176, HPeV5-552322, HPeV6-550389 та два клінічних ізоляти HPeV3 були отримані в лабораторії клінічної вірусології Академічного медичного центру, Амстердам, Нідерланди. Для запасів вірусу HPeV1, HPeV2, HPeV3 A308/99 (подарунковий тип Hiroyuki Shimizu і Miyabe Ito) та HPeV4-6 культивували в клітинній лінії HT29 в мінімальному незамінному середовищі Eagle з L-глутаміновою кислотою (0, 2 ×), неісенціальні амінокислоти (1x), стрептоміцин (0,1 мкг мл -1), пеніцилін (0,1 мкг мл -1) та 2% інактивований FBS (клітини HT29 підтримували в середовищі, що містить 8% інактивованого тепла FBS). Ці штами використовувались в аналізах зв'язування та нейтралізації антитіл AT12-015.
Вироблення антитіл та отримання фрагментів Fab.
Моноклональне антитіло людини AT12-015 проти ізоляту HPeV3 152037 готували з крові людини 15. Коротко кажучи, клітини CD27 + IgG + B людської пам’яті культивували методом AIMSelect у здорових донорів, які одужали від ізольованої 152037 інфекції HPeV3, підтвердженої 1 роком раніше. Поодинокі клітини субклонували з культур клітин В, в яких супернатант продемонстрував зв'язування з інфікованими HPeV3 клітинами за допомогою імунофлюоресценції. РНК виділяли з моноклональних В-клітин для відновлення важких та легких ланцюгових послідовностей антитіла. Унікальні послідовності використовували для генерування рекомбінантного білка в клітинах 293 Т. Антитіла IgG1 додатково очищали за допомогою колонок HiTrap з білком А на приладі ÄKTA (GE). Фрагменти AT12-015 Fab отримували з використанням мікропрепарування Pierce Fab згідно з інструкціями виробника для досягнення повного перетравлення антитіла. Отримані фрагменти Fab змішували з капсидами HPeV3 у молярному співвідношенні 60 до 1 та інкубували протягом 30 хвилин при 37 ° C у 1 х TNM, щоб дати змогу утворитись вірусно-Fab-комплексу перед склуванням.
Аналіз нейтралізації HPeV
Нейтралізацію ізоляту HPeV3 152037 AT12-015 тестували шляхом зараження клітин Vero попередньо інкубованими вірусами (100 одиниць TCID50) з різною кількістю антитіла (0,03–7,5 мкг мл -1) при 37 ° С протягом 1 години. За зараженими клітинами спостерігали за появою CPE кожні 24 год протягом 7 днів. На 7 день РНК виділяли з супернатанту за допомогою набору для виділення загальної нуклеїнової кислоти за допомогою приладу MagnaPure LC (Roche Diagnostics), зворотної транскрипції та кДНК (комплементарна ДНК) використовували для оцінки кількості вірусної копії за допомогою ПЛР у реальному часі Прилад LC480 (діагностика Roche) 16. Здатність AT12-015 нейтралізувати ізолят HPeV3 152037 також тестували на BGM (нирка мавп зеленого буйвола), A549 (аденокарцинома товстої кишки людини) та Caco2 (аденокарцинома товстої кишки). Крім того, перехресну нейтралізацію AT12-015 проти HPeV1, HPeV2, HPeV4, HPeV5 та HPeV6 також тестували в клітинах Vero. Імунофлуоресценція клітин, інфікованих HPeV1-6, також перевірялася за допомогою AT12-015.
Кріоелектронна мікроскопія та обробка зображень.
Інактивований формальдегідом очищений HPeV3 вітрифікували, наносячи 3 мкл зразка на сітки Quantifoil R3.5/1, сушили протягом 2 с при відносній вологості 92% і занурювали в рідкий етан за допомогою Leica EM GP. Решітки досліджували на просвічувальному електронному мікроскопі FEI Titan Krios з поправкою на Cs при 300 кэВ. Зображення реєстрували на детекторі Falcon II в умовах низьких доз з номінальним збільшенням 59000 × при розмірі вибірки 1,14 Å на піксель. Сім кадрів на зображення було зібрано в режимі підрахунку за допомогою автоматизованого програмного забезпечення для збору одиничних частинок FEI EPU. Плівкові кадри спочатку вирівнювались між собою за допомогою програмного забезпечення motioncorr 17 до подальшої обробки.
Для асиметричної реконструкції орієнтація найкращих 41 845 частинок була уточнена протягом додаткових 16 раундів за допомогою 3-мірної автоматичної опції уточнення в Relion, не накладаючи жодної симетрії. Остаточне уточнення між двома незалежними наборами даних дозволило 10,36 Å на основі критерію FSC 0,143.
Аликвоти комплексів HPeV3-Fab AT12-015 вітрифікували на теплорозвантажувальній надтонкій вуглецевій плівці, що містить мідь та вуглецеві сітки (TED PELLA), використовуючи саморобну гільйотину. Решітки досліджували на просвічувальному електронному мікроскопі FEI Tecnai F20 при 200 кэВ з використанням кріостата Gatan 626. Зображення записували на плівки Kodak SO-163 в умовах низьких доз з номінальним збільшенням 50 000 ×. Плівки оцифровували на сканері Zeiss (Photoscan) із розміром кроку 7 мкм, даючи розмір пікселів 1,4 Å на піксель. Функцію передачі контрасту кожної мікрофотографії оцінювали, як описано вище. Частинки збирали на мікрофотографії з інтервалом 4 × за допомогою програми RobEM 21 із розміром коробки 101 піксель. Модель випадкового старту була створена із 150 частинок за допомогою AUTO3DEM v 4.05.1 (посилання 21). Повний набір даних був розділений на дві частини, а орієнтації та походження визначались на основі незалежних реконструкцій, створених кожним набором. Кінцева роздільна здатність 15 Å 564 частинок була досягнута на основі критерію 0,143 FSC.
Побудова моделі
Структури трьох капсидних білків HPeV3 були передбачені порівняльним моделюванням декількох шаблонів із використанням сервера I-TASSER 25. Структури шаблонів для VP0 включали визначений користувачем шаблон, що містить злиту структуру терміналу людського ентеровірусу VP4 (PDB ID: 3vbf) N для змивання ентеровірусу людини VP2 (PDB ID: 3vbo) 26, а також хвороби ящуру. вірус (PDB ID: 1qqp) 27, поліовірус 1 (PDB ID: 1pov) 28, бичий ентеровірус (PDB ID: 1bev) 29, еховірус 1 (PDB ID: 1ev1) (посилання 30) та гепатит A (PDB ID: 4qpg ) 31. Оцінка С для найкращої моделі становила –1,03.
Для VP3 був використаний порожній шаблон, наданий користувачем ентеровірусом (PDB ID: 3vbo) 26, на додаток до таких шаблонів, вибраних програмою: coxsackievirus A9 (PDB ID: 1d4m) 32, echovirus 1 (PDB ID: 1d4m) PDB: 1ev1) (посилання 30), ентеровірус людини (PDB ID: 3vbf) 26, ентеровірус людини (PDB ID: 3vbh) 26, вірус коксакі людини A16 (PDB ID: 4jgy) 7. Оцінка C для найкращої моделі становила –0,61.
Для VP1 порожній людський ентеровірус (PDB ID: 3vbo) 26 був наданий як зовнішній шаблон на додаток до таких шаблонів, вибраних програмою: людський ентеровірус 71 (PDB ID: 3vbh) 26, людський ентеровірус 71 (PDB ID: 3zfe) 33, вірус коксакі людини A16 (ідентифікатор PDB: 4jgy) 7, ентеровірус людини 71 (ідентифікатор PDB: 4cdq) 34, вірус коксакі A 9 (ідентифікатор PDB: 1d4m) 32, риновірус людини 14 (ідентифікатор PDB: 1ncq) 35. Оцінка C для найкращої моделі становила –0,75.
Моделі гомології були жорстко встановлені на карті HPeV3, використовуючи функцію "придатність до карти" в UCSF-Chimera 36. За допомогою функції зонування в UCSF-Chimera 36 карту капсидів HPeV3 розділили на асиметричну одиницю радіусом 6 Å, використовуючи модель VP0-VP3-VP1 з жорсткою посадкою. Модель VP0-VP3-VP1 була гнучко вписана в асиметричний блок, використовуючи порівняння результатів двох різних програм гнучкої підгонки iMODfit 37 та FlexEM 38, щоб досягти консенсусної підгонки 39. Обидві гнучкі програми регулювання використовувались із налаштуваннями за замовчуванням. Потім візерунки допрацьовували вручну в Coot 40 шляхом обрізання візерунків там, де щільність не була очевидною, поліпшення прилягання хребта та регулювання важких бічних ланцюгів залишків фенілаланіну, тирозину, триптофану та аргініну там, де їх видно. Моделі були вдосконалені, вдосконаливши фактичний простір у Phenix 41. Всі візуалізації проводились у UCSF-Химера 36 .
Одну копію структурованої області РНК було вилучено за допомогою 'Bulk Eraser' в UCSF-Chimera 36. Вилучений об’єм міг містити близько 30 нуклеотидів. 30-нуклеотидна область (7181-7210) на 3 'неперекладеному кінці ізольованого геному HPeV3 152037 (ідентифікатор GenBank: GQ183026) була складена на веб-сервері RNAfold 42 і змодельована у RNAComposer 43. Ця модель РНК була пристосована до витягнутого об'єму за допомогою опції "Припасувати до карти" в UCSF-Chimera 36 .
Слід AT12-015 був оцінений шляхом накладання атомної моделі HPeV3 на реконструкцію комплексу HPeV3-Fab AT12-015 та створення дорожньої карти для атомної моделі з легендарним слідом як контурними лініями в RIVEM 44. Всі амінокислотні залишки, виявлені на поверхні, у цьому сліді були взяті як епітопи для Fab.
Додаткова інформація
Коди доступу: Карти щільності ікосаедрично-симетричних HPeV3, асиметричних HPeV3 та HPeV3-Fab AT12-015 були депоновані в Банку даних електронної мікроскопії під кодами доступу EMD-3137, EMD-3322 та EMD-3138, відповідно. Моделі, скориговані на HPeV3, були депоновані в банку даних про білки в Європі під ідентифікатором PDB: 5APM. Всі вихідні дані, зібрані для реконструкції HPeV3, доступні через файл пілотного зображення електронної мікроскопії з кодом доступу EMPIAR-10033.
Як цитувати цю статтю: Shakeel, S. та співавт. Множинні взаємодії, що стабілізують капсиди, виявлені в структурі високої роздільної здатності нового пікорнавірусу, що спричиняє неонатальний сепсис. Нац. Комун. 7: 11387 doi: 10.1038/ncomms11387 (2016).
- Папайя, фрукт з багатьма користями для здоров'я, який ви повинні включити у свій щоденний раціон -
- Множинні та корисні властивості сперми Placeres S Moda EL PAÍS
- Що є ідеальним споживанням калорій для людини висотою 5 футів і 6 дюймів вагою 95 кг?
- Жінкам, які хочуть схуднути, слід харчуватися дієтою з високим вмістом молочного білка -
- Дев'ять фруктів з численними перевагами для шкіри